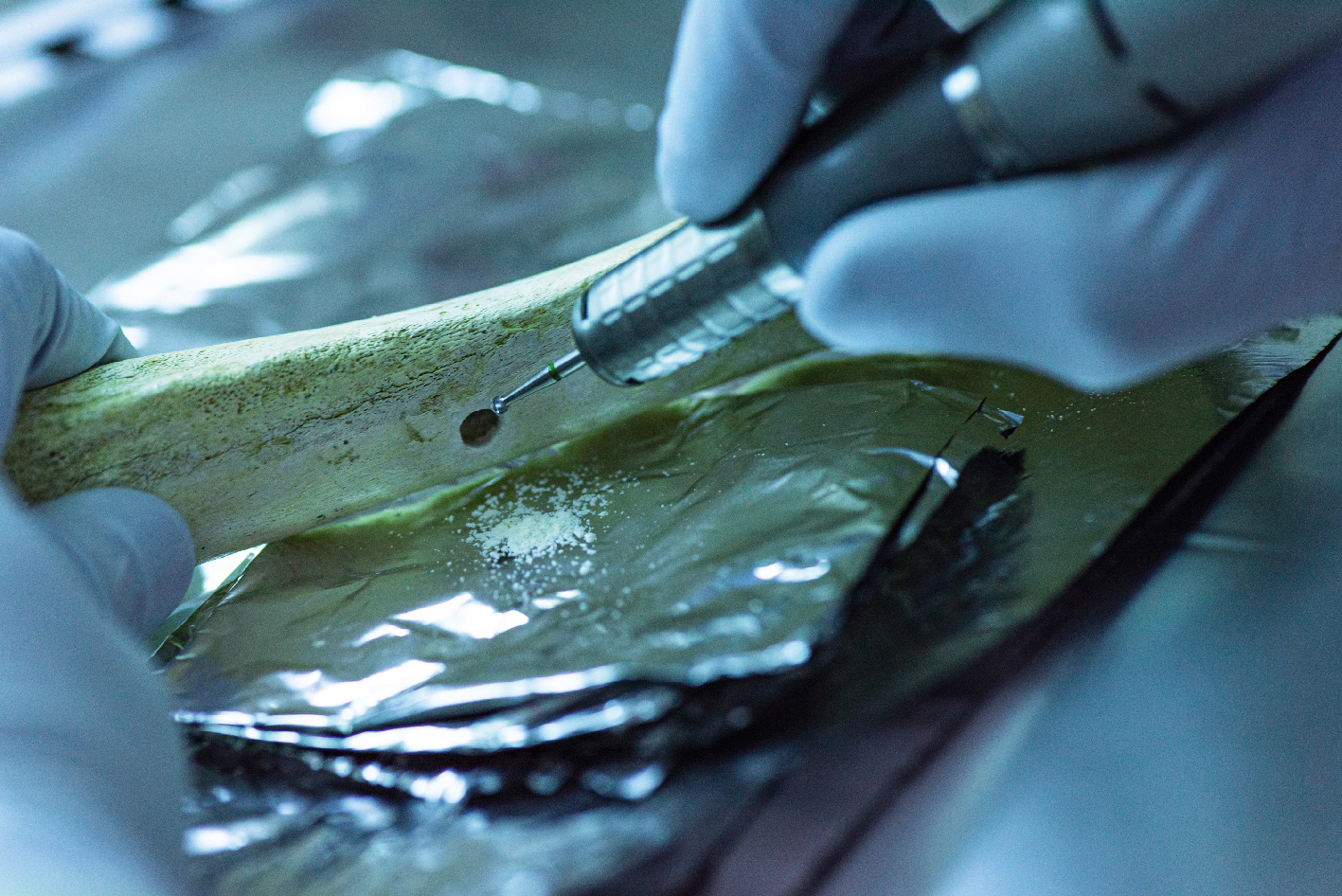
Aufbereitung einer Knochenprobe: „Alte DNA“ zu gewinnen, gelingt nur in extrem kontrollierten Laborumgebungen. Foto: Max-Planck-Institut für Evolutionäre Anthropologie
Es gib wissenschaftliche Begriffe, die es in die Alltagswelt geschafft haben. LMU-Wissenschaftler erklären an dieser Stelle solche Ausdrücke – nicht nur mit einer reinen Definition, sondern auch mit einer kurzen Geschichte ihrer Popularität.
„Stirbt ein Organismus, wird seine DNA in seinem Körper abgebaut, wie die übrigen Biomoleküle auch. Die DNA ist jedoch ein sehr widerstandsfähiges Molekül. In Haaren, Knochen oder bei Pflanzen in Samen kann sie über Tausende von Jahren erhalten bleiben. In den letzten 40 Jahren haben Forscher neue Labortechniken entwickelt, um diese ,alten‘, stark degradierten DNA-Moleküle aus paläontologischem und archäologischem Material zu extrahieren. Mit Erfolg: Jüngst hat der Genetiker Svante Pääbo den Medizinnobelpreis erhalten, weil er mit ihrer Hilfe das Erbgut längst ausgestorbener Frühmenschen rekonstruierte.
Die Arbeit mit alter DNA ist schwieriger als mit ,moderner‘ DNA, da die DNA-Menge in alten Proben oft sehr gering ist. Alte DNA erfordert eine besondere Behandlung und ein Arbeiten in extrem kontrollierten Laborumgebungen, um DNA-Verunreinigungen durch Personen, die mit der Probe umgehen, oder durch die Umwelt zu vermeiden.
Biotechnologische Fortschritte haben die Arbeit mit diesen winzigen Mengen an DNA-Molekülen möglich gemacht. Der vielleicht wichtigste ist die Polymerase-Kettenreaktion (PCR), ein Verfahren zur Vervielfältigung von DNA-Molekülen. Es erlaubt, DNA-Moleküle in ausreichender Menge zu erzeugen, um sie zu sequenzieren. Neue Sequenzier-Technologien erlauben, ganze Genome in einem Tempo zu sequenzieren, das noch vor 15 Jahren für unmöglich gehalten wurde.
Der Zugang zu genetischen Informationen aus der Vergangenheit hat Bereiche wie Paläontologie, Paläoanthropologie und Archäologie revolutioniert. Durch die Entschlüsselung des DNA-Codes alter Organismen, darunter auch von Mensch oder Neandertaler, haben Forscher nun Zugang zu einer Art Zeitmaschine. Sie macht Momentaufnahmen aus der fernen Vergangenheit möglich. So können Genetiker nun die Geschichte unserer Spezies in noch nie dagewesener Detailtiefe erforschen und etwa untersuchen, was uns von unseren entfernten Vettern, den Neandertalern, unterscheidet. Oder verstehen, wie wir die Welt kolonisiert haben, uns an Erreger wie die Schwarze Pest angepasst haben, aber auch, wie und wann wir Pflanzen wie Reis, Weizen und Gerste domestiziert haben und Tiere wie Hunde oder Pferde unsere Begleiter wurden.
Anhand von Genominformationen aus vergangenen Ökosystemen, auch aus ausgestorbenen und noch lebenden Arten wie Mammuts, Elefanten und Wölfen, untersuchen Forscher, wie Arten während der großen klimatischen Veränderungen der letzten Million Jahre überlebten und sich anpassten oder ausstarben. Damit hat ,alte DNA‘ ein enormes Potenzial, uns bei Aussagen zur Zukunft zu helfen, dazu, wie wilde aber auch domestizierte Arten auf die Klimakrise reagieren werden.“
Protokoll: huf
Prof. Dr. Laurent Frantz ist Professor für Paläogenomik der Haustiere an der Tierärztlichen Fakultät der LMU.





















0 Kommentare